一、技术原理
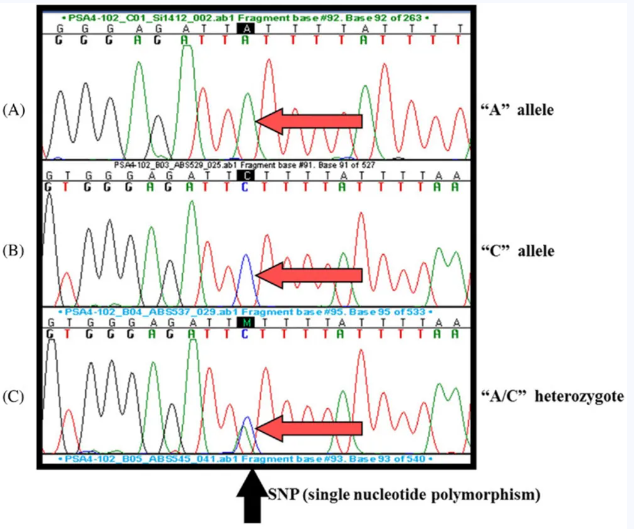
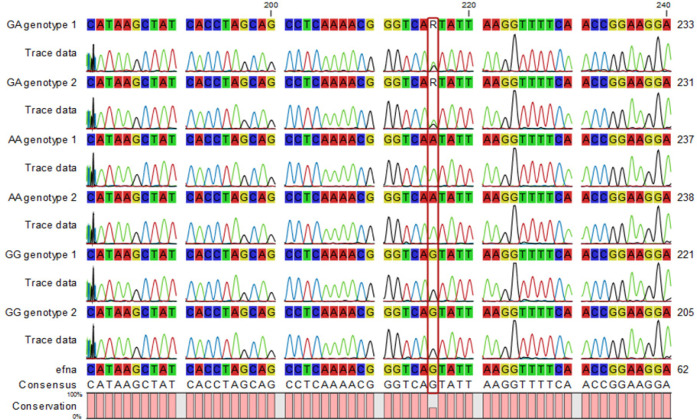
Sanger测序原理:使用DNA聚合酶和带有荧光标记的双脱氧核苷酸(ddNTP),ddNTP缺少3'-OH基团,能够终止DNA链的延伸。通过区分不同ddNTP的荧光标记,可以确定DNA链的序列。
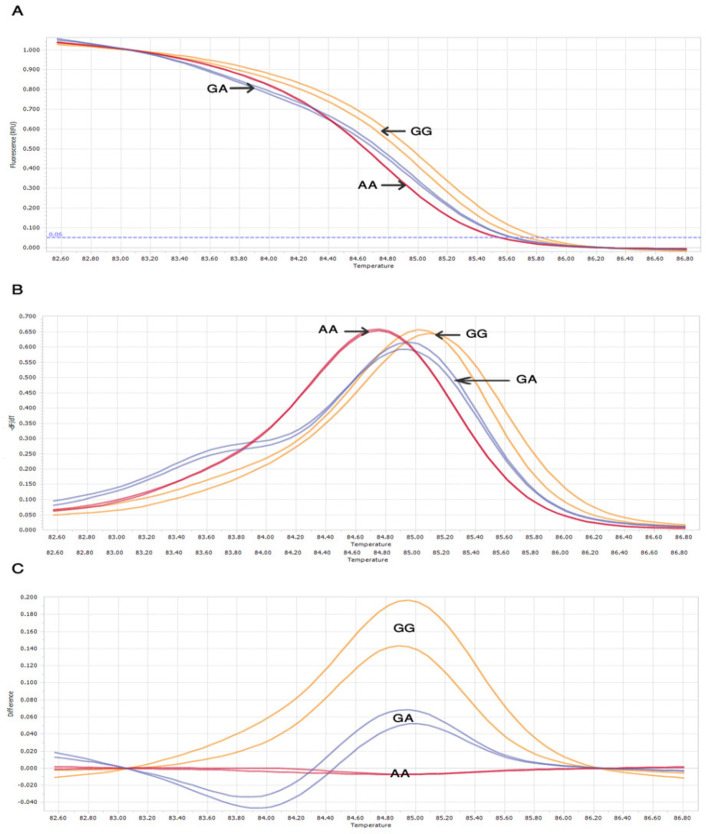
TaqMan探针法原理: TaqMan探针5'端和3'端分别标记有报告荧光基团和淬灭基团。在PCR反应过程中,如果探针与模板DNA完全匹配,Taq聚合酶会将探针切割,使得荧光基团与淬灭基团分离,从而发出荧光信号。根据荧光信号的强度变化,可以判断样本的基因型。
SNaPshot法原理: 使用测序酶和荧光标记的ddNTP,通过延伸引物与PCR产物模板的反应,引物延伸一个碱基即终止。通过测序仪检测峰的移动位置和颜色,确定SNP位点和碱基种类,从而确定样本的基因型。
基因芯片法原理: 基因芯片法通过将大量已知序列的DNA探针固定在芯片上,然后与标记的样品DNA进行杂交。通过检测每个探针的杂交信号强度,可以获取样品DNA的序列信息。
质谱法检测SNP的原理:通过PCR扩增后的产物,加入特异延伸引物,在SNP位点上延伸1个碱基。然后将样品与芯片基质共结晶,通过质谱仪检测,根据质荷比分离并检测不同的分子质量,从而进行SNP分型。
二、实验流程
Step1:DNA提取
Step2:PCR扩增目标区域
Step3:SNP分型(芯片法、测序法等)
Step4:结果分析
三、技术总结
可根据需要选择不同的检测方法,尽量首选成本低、样品制备过程简单的方法;
Sanger测序等方法结果易受到干扰,导致数据不清晰,再考虑使用质谱等技术。
四、样本要如何准备?
细胞样本:细胞数1×10^7个,可以是细胞液或细胞沉淀,干冰运输;
组织样本:组织量≥100 mg,干冰运输;
血液样本:样品需使用EDTA抗凝,体积≥2.5 mL,低温运输,长时间运输需要干冰;
FFPE样本:FFPE 组织块低温运输;
PCR产物:DNA:体积≥20 μL,浓度≥50 ng/μL,低温运输。